PyMOL:安装及入门笔记
PyMOL 是一款基于 Python 开发的三维结构可视化的软件。个人觉得同 VMD 及 SPDBV 相比,其操作更加简单灵活、图形更加美观,而且可以使用命令行进行批处理,本文主要记录鼠标操作的相关内容。
1.安装
由于 PyMOL 是基于 Python 开发的,所以需先安装 pmw(Python 可以通过直接安装来 Anoconda 完成)。其还依赖 Pmw ,可以通过 pip install pmw 提前安装好,若网络不好,可通过更改 pip 的镜像地址来改善,如使用清华源来安装 pip install pmw -i https://pypi.tuna.tsinghua.edu.cn/simple。
随后从下方链接中下载 pymol,其版本比较多所以在下载的时候需要根据系统及 Python 版本来进行选择,我的电脑是 Windows 64 位系统,Python 版本是 3.7,所以选择下载 pymol-2.5.0a0-cp37-cp37m-win_amd64.whl,然后在cmd 中运行 pip install pymol-2.5.0a0-cp37-cp37m-win_amd64.whl 来安装,故后续记录均针对该版本,老版本不做说明。
下载地址:https://www.lfd.uci.edu/~gohlke/pythonlibs/
下载地址中不仅包含 pymol,还有pmw,所以 pmw 同样可以使用如上方法本地安装,避免因网络问题而失败的情况。
安装完成后,启动图标位于 C:\ProgramData\Anaconda3\Scripts\pymol.exe ,双击即可启动,建议发送到桌面快捷方式或固定到开始屏幕。也可以将 bdb 文件的默认打开方式设置为 PyMOL。
2.入门笔记
2.1 主界面介绍
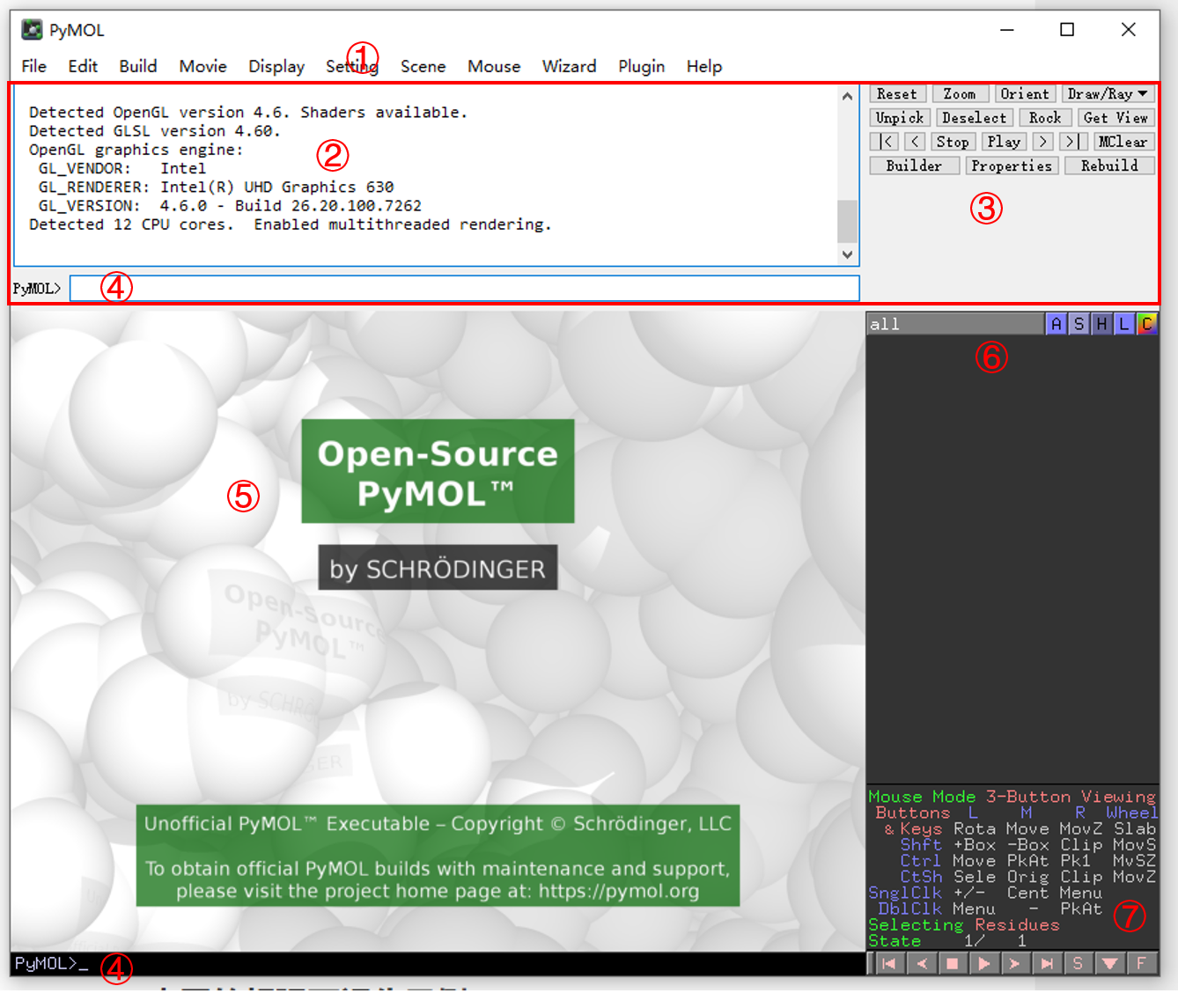
PyMOL 的主界面如下,其中红框部分为 External GUI,可以点击 Display - External GUI - Toggle Floating 或使用快捷键 Ctrl + E 将该部分与初始界面分离或合并。External GUI 主要有三个功能模块:② 操作记录窗口、③ 常用命令窗口和 ④ 命令输入窗口,余下模块分别如下:
- ① 菜单栏
- ⑤ 可视化窗口
- ⑥ 对象窗口,显示编辑的文件,类似于图层的概念。
- ⑦ 模式窗口,主要控制鼠标的控制模式
2.2 切换工作路径及载入文件
可以通过点击 File - Working Directory - Change... 来切换路径,也可以输入指令进行切换,建议不同项目使用不同的工作路径。
载入文件有三种方式:
- 点击
File-Open...加载文件 - 拖拽文件至可视化窗口
- 使用
load指令
# 切换至 C:\Users\DELL\Desktop\pymol\pdb
cd C:\Users\DELL\Desktop\pymol\pdb
# 查看当前工作路径
pwd
# 加载 protein.pdb
load protein.pdb
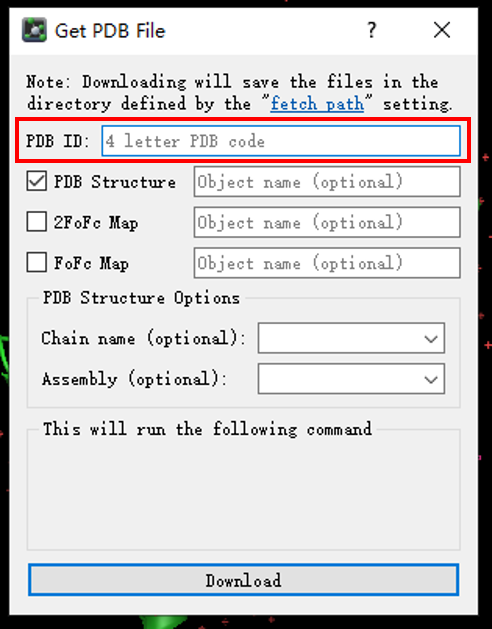
PyMOL 可以通过 File - Get PDB... 来下载 PDB 文件,如下图所示,只需在红框中填入 四字母的 PDB ID(如 4hbk),点击下方的 Download 即可。
2.3 简单的平移、缩放
- 平移:按住鼠标中键不放后移动鼠标
- 旋转:按住鼠标左键不放后移动鼠标
- 缩放:按住鼠标右键不放后移动鼠标
- 切割:滚动鼠标中键
若鼠标功能与上述不符,可以点击 File - Reinitialize - Original Settings 来恢复默认设置。
2.4 设置背景颜色及显示结构的序列
设置背景颜色:Display - Background - white
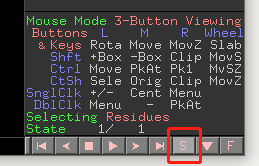
显示结构序列:Display - Sequence 或者直接点击模式窗口中的 S,如下图红框部分所示。
2.5 对象窗口
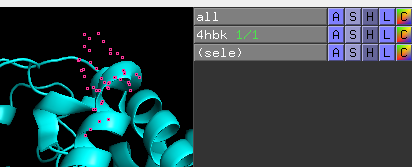
载入 4hbk 蛋白后,使用鼠标随意选择几个氨基酸,对象窗口会出现三个object,其中 all 代表的是其中包含的所有项目,4hbk 则是刚刚载入的蛋白,而(sele)就是随意选择的几个氨基酸。
每个项目中后面均有A、S、H、L 和 C 五个选项,意义如下:
- A: Action,包含放大、剧中,复制,显示及比对等一系列的操作。
- S: Show,设置不同的显示方式。
- H: Hide,隐藏显示的方式,与 S 基本对应。
- L: Lable,设置不同的标记方式。
- C: Color,设置不同的着色方式。
2.5.1 Show 和 Hide
需要注意 Show 中有如下两种改变外观的方式,以变化为 licorice 为例:
S-licorice:在原有的基础上,添加 licorice 这一层外观S-as-licorice:改变外观为 licorice
了解了有无 as 的区别后,根据需求更改为所需样式即可。Hide 基本和 Show 基本对应,故在此不一一赘述。
2.5.2 Action
Action 中的包含如下所示较为多样的功能,需注意其中标红的为不可逆的操作,不能通过 Ctrl + Z 恢复。
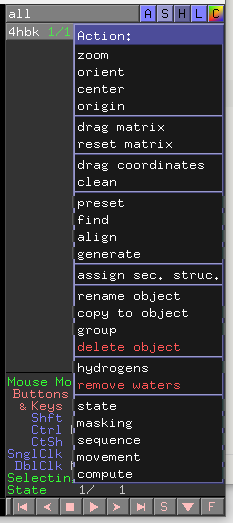
2.5.2.1 Action 中显示操作
Action 中 preset 可以便捷的修改显示方式,主要有以下几种显示方式:
- simple:简单形式
- ball and stick:球棍模型
- b-factor putty:基于 b-factor 数值显示蛋白的柔性
- pretty:美观的 cartoon 样式
- publication:高质量出版标准
preset 中还包含查看小分子和蛋白的氢键作用的功能,具体操作如下,其中 ligand sites 后有许多显示选项,根据需求选择。下图展示 cartoon 选项的结果,红色的小分子和蛋白作用的结果展示。
A-preset-ligand sites
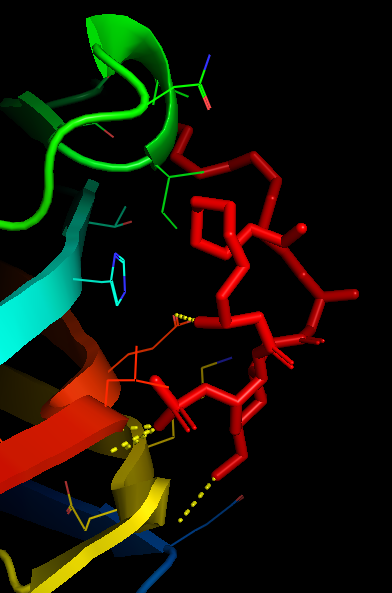
2.5.2.2 Action 中对象的复制、剪切、删除和重命名
- 复制:
A-Copy to object-new - 重命名:
A-rename object - 删除:
A-delete object - 剪切:选中需要剪切的部分后点击
A-Extract
2.5.2.3 Action 中氢原子和水分子的相关操作
水分子只有标红的删除操作:A - remove waters
氢原子在 line 和 stick 模式下面可见,以 4hbk 为例,先将模式调整为 S - as - licorice ,再依次进行下面操作并体会变化。
A-Hydrogens-add:增加所有氢原子A-Hydrogens-remove non polar:删除所有非极性氢原子,可以看到C上的氢原子全部被删除A-Hydrogens-remove:再次删除所有氢原子A-Hydrogens-add polar:增加极性氢原子
2.5.2.4 Action 显示蛋白的静电势图
操作和结果如下所示:
Action - generate - vacuum_electrostatics - protein contact potentia
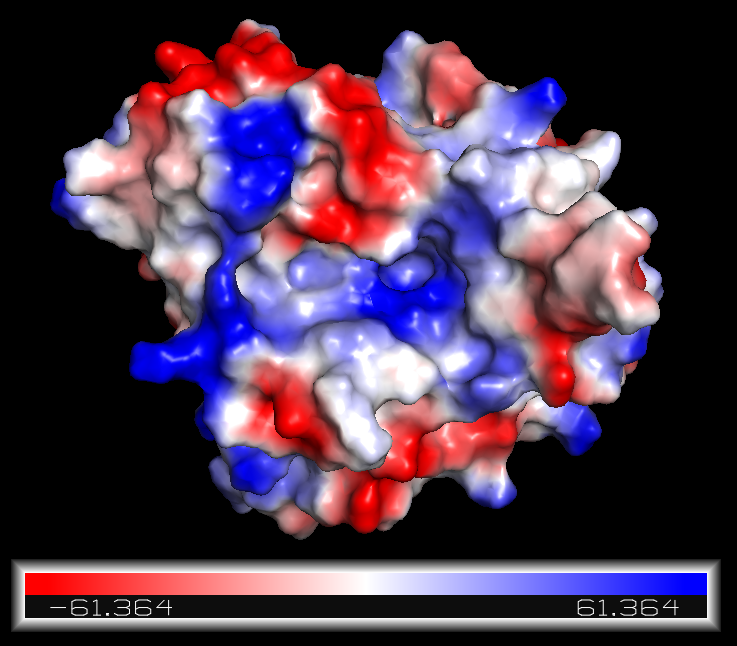
2.5.3 Color
C-by element:按原子类型着色C-by chain:按链着色C-by ss:按二级结构着色C-reds:整体标红
2.5.4 Lable
选择好需要 Lable 的对象后,可以对其进行如下不同方式的标记。
L-residue:在 α 碳原子上标记其氨基酸残基三字母名字和编号L-residue(oneletter):标记其氨基酸残基一字母名字和编号L-one letter code:标记其氨基酸残基一字母名字L-element symbol:显示对象上所有原子的元素名字L-vdw radius:显示原子的范德华半径L-clear:删除该对象上所有的 Label
由于后续会调整不同的角度,所以默认 Lable 位置很大概率需要调整,调整步骤如下:
- 单击下图模式窗口中红框部分,将 Mouse Mode 由 Viewing 改为 Editing。
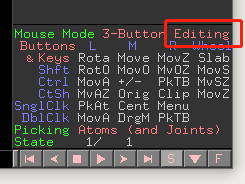
- 按住
Ctrl后使用鼠标左键拖拽 Label 至适当的位置,拖拽过程中鼠标左键同样是按住不放的状态,调整结束后别忘了将 Mouse Mode 改回 Viewing 状态。
2.6 测量距离
- 点击
Wizard-Measurement,如下图所示在界面右下方会出现 Measurement 界面。 - 选择所需测量距离的两个原子,如有多个需要测量,成对选择即可。
- 测量完成后,点击 done 结束。
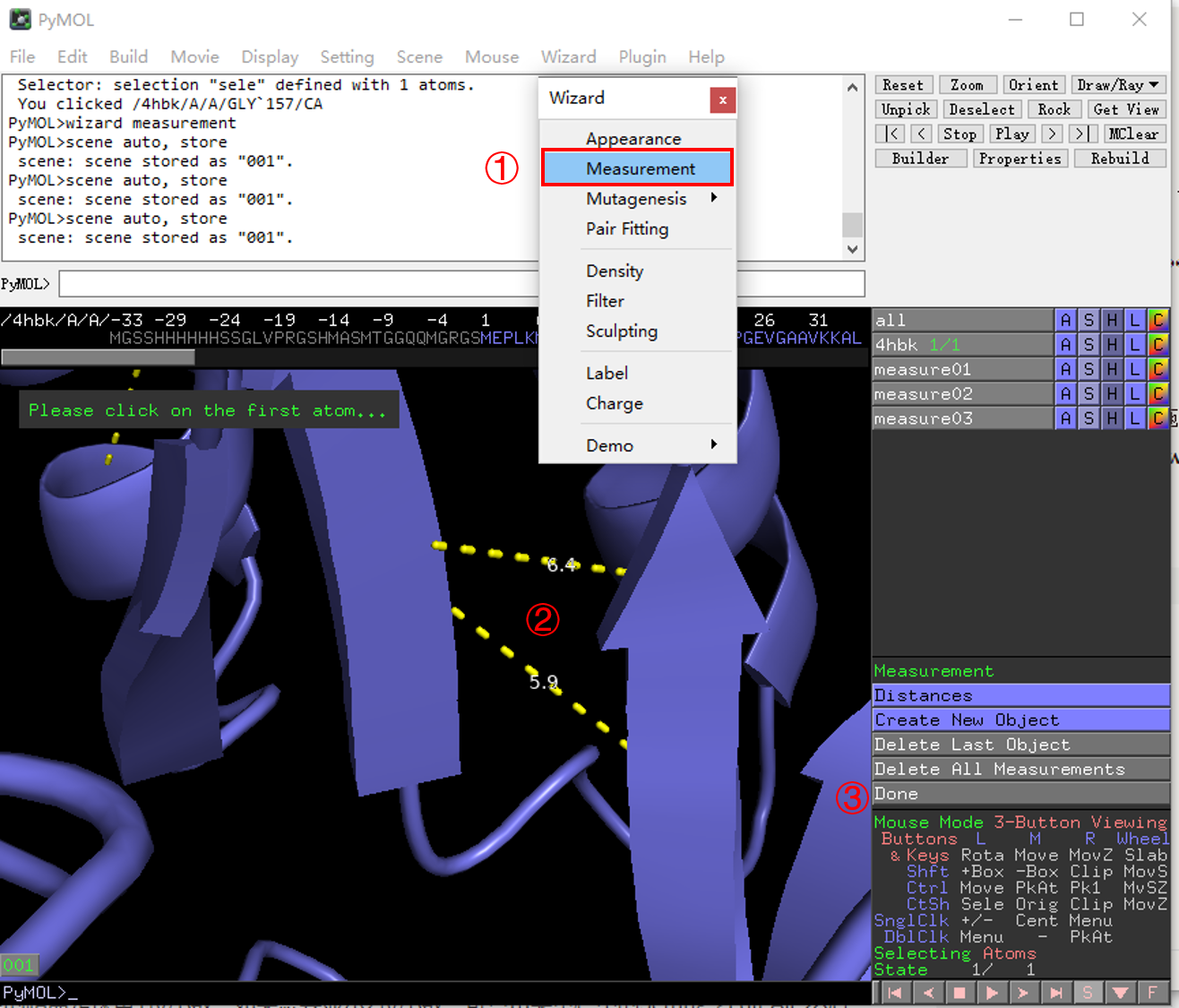
注意:默认测定的距离是保留 1 位小数,如果想要显示 2 位小数,可点击菜单栏中的 Setting - Edit All,然后搜索 label_digits,把后面的数值设置为2就可以了。
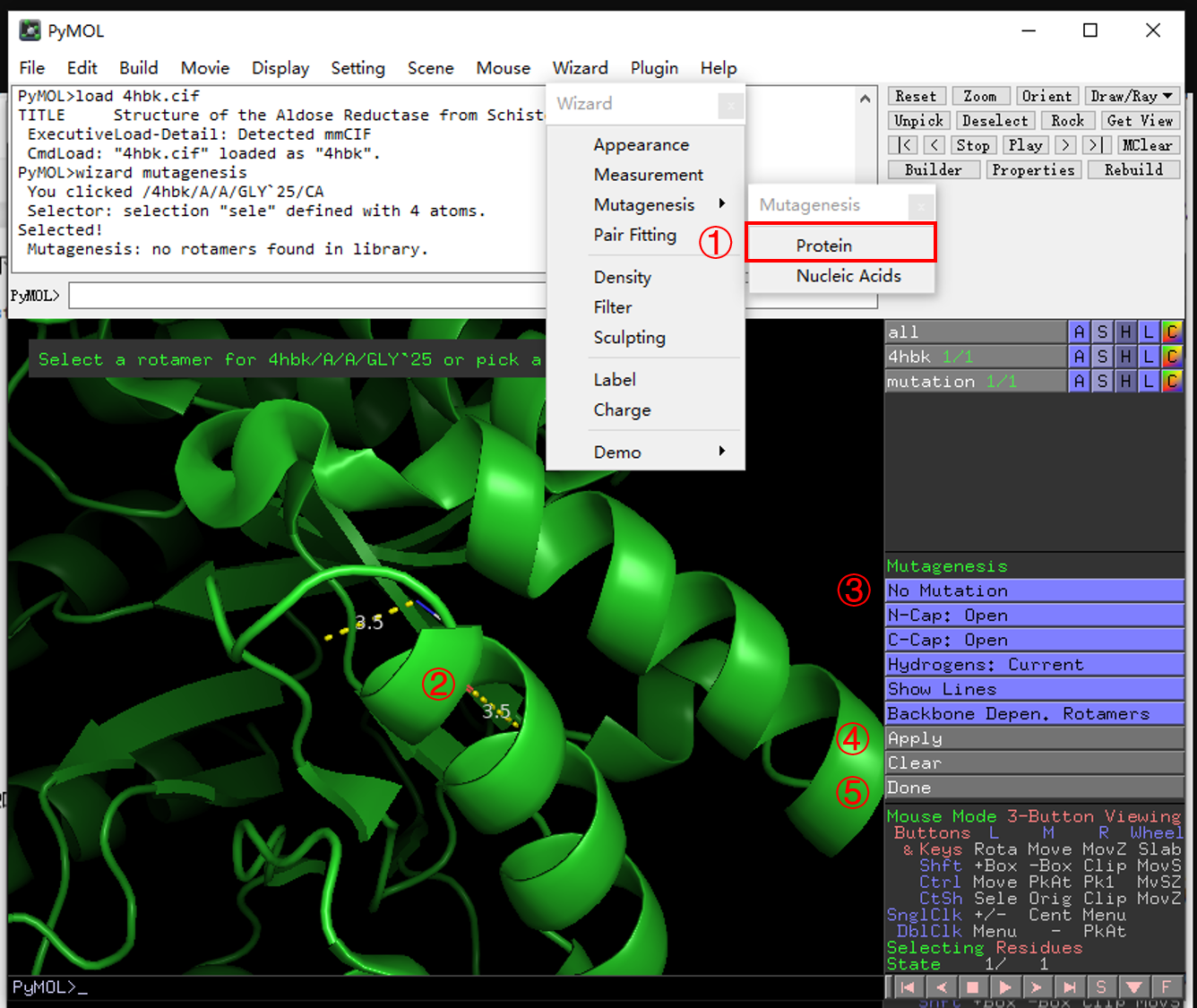
2.7 突变氨基酸残基
- 在菜单中点击
wizard-mutagenesis-protein,如下图所示打开氨基酸突变栏。 - 选择所需突变的氨基酸。
- 单击
No Mutation后选择所需突变成的氨基酸。 - 单击
Apply将氨基酸替换为目的氨基酸。 - 单击
Done完成突变。
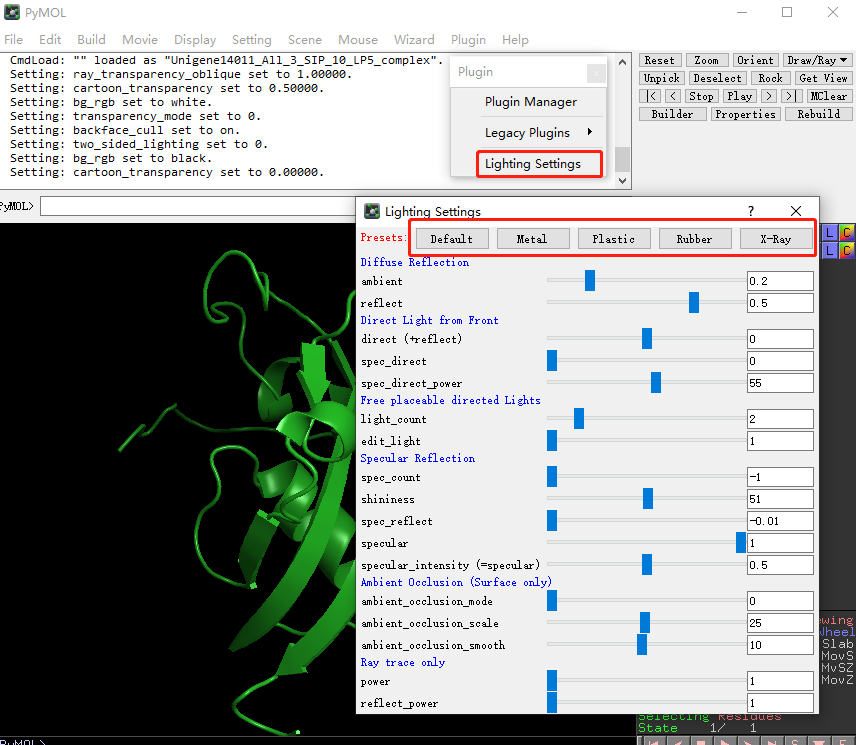
2.8 设置透明度和光照模式
透明度设置位于 Setting 中的 Transparency。其中不仅可以设置不同模式的透明度(如cartoon、surface 和stick等),还预设有4种不同的透明方式,可以自行修改观察效果。
光照模式位于 Plugin 中的 lighting Settings 中。里面预设 5 种类型(default, metal, plastic, rubber and X-ray),根据需求自行修改即可。
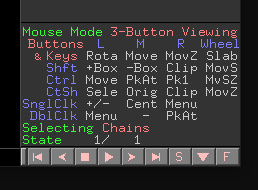
2.9 鼠标的选择模式
如下图所示,简单介绍 Mouse Mode 为 Viewing 情况下的 Selecting 模式,有如下几种:
- Residues 残基选择模式
- Chain 链选择模式
- Segments 片段模式
- Objects 模式
- Molecules 分子选择模式
- C-alphas α碳原子模式
- Atoms 原子选择模式
2.10 保存项目及导出结果
File-Save Session:保存为pse 文件,方便修改调整。File-export image as-png:保存图片,可以使用如下图所示的Draw/Ray按钮,在设置图片大小及分辨率后保存图片。File-export molecule:然后从selection的下拉框中选择需要导出的object,all 代表所有的object。
参考资料:
1.PyMOL中文教程
2.免费开源版(非Edu版)PyMOL安装教程
3.win10系统下如何安装pymol?
4.PyMOL | Pymol绘图教程(一)
5.高质量PyMOL作图教程
6.Pymol(1.8.6)作图技巧之cartoon和surface镶嵌模型
7.教程:Pymol, Chimera预测点突变蛋白结构
