PyMOL:命令行操作学习笔记
本文主要记录 PyMOL 中的一些命令行操作,PyMOL 的主界面虽然有两个输入指令的窗口,但个人认为 External GUI 里面的指令输入框更为方便,不仅能查看输入的记录,也能在分离出主界面后放大并置于合适的位置进行命令行操作。另外 PyMOL 自带 tab 键补齐的功能,所以 PyMOL 对命令行操作还是比较友好的。
1.查看指令的帮助文档(help and ?)
下面两种方式都可以查看指令的帮助文档,区别是前者比后者给出的信息更加全面。
help command
command ?
PyMOL 的所有指令都可以通过点击 Help - PyMOL Command Reference 在如下网址中显示。
PyMOL Command Reference: http://pymol.org/pymol-command-ref.html
2.查看和切换工作路径(pwd、cd and ls)
pwd:查看当前路径
cd C:/Users/DELL/Desktop/pymol/test:切换到 test 文件夹中
ls:查看当前路径中的文件
注意:Windows 系统中默认的路径中的反斜杠可以使用 / 来替换。
3.下载和载入蛋白结构(fetch and load)
使用 fetch ID 下载蛋白结构。默认从 pdb 网站下载,并且下载完成后自动载入。
fetch 6MVD # 下载格式为cif的6MVD的结构
fetch 6MVD, type=pdb # 下载格6MVD的结构,但格式指定为pdb
fetch 6MVD, type=pdb, name="6MVD-test" # 下载格式为pdb的6MVD的结构,但命名为 6MVD-test
使用 load filename 载入蛋白结构,其后也可以使用结构对应的 url 来下载并载入,所以其同 fetch 的差别在于不能根据 ID 搜索。
load http://files.rcsb.org/download/6MVD.pdb # 从URL下载6MVD后载入
load 6MVD.pdb # 载入存在于当前目录下的6MVD.pd
注意:虽然两个指令都可以下载结构文件,但是不建议使用 PyMOL 来下载。
4.object 的排序、隐藏、保存和删除(order、disable、enable、delete、save and remove)
依次载入 6mvd 和 4hbk 后进行如下操作,同时主要观察对象和显示窗口中 object 的变化:
order *,yes # 根据 object 名字进行排序
order 6mvd 4hbk # 根据设定的顺序排序
disable 6mvd # 隐藏 6mvd
enable 6mvd # 显示 6mvd
delete 6mvd # 删除 6mvd
remove resn hoh # 删除水分子,由于并没有删除水分子的选项,此处是基于残基匹配水分子来删除的
save 4hbk.cif # 保存 4hbk
disable all # 删除所有object
load 4hbk.cif # 载入 4hbk 查看水分子
注意:载入多个结构时,由于缩放问题,显示窗口只会显示最后一个载入的结构,这时使用 reset 或 orient 就可以显示载入的所有结构。
5.保存图片相关设置(png、ray and bg_color)
使用 png 来输出 png 格式的图片,有如下可控参数。
png filename [, width [, height [, dpi [, ray]]]]
filename:输出图片的名字
width 和 height:提供 pixels(默认), inches (in) 和 centimeters (cm)三种单位
dpi:dots-per-inch
ray:0 或 1,默认为 0,表示不使用,当设置为 1 时,之前需运行 ray 渲染光线
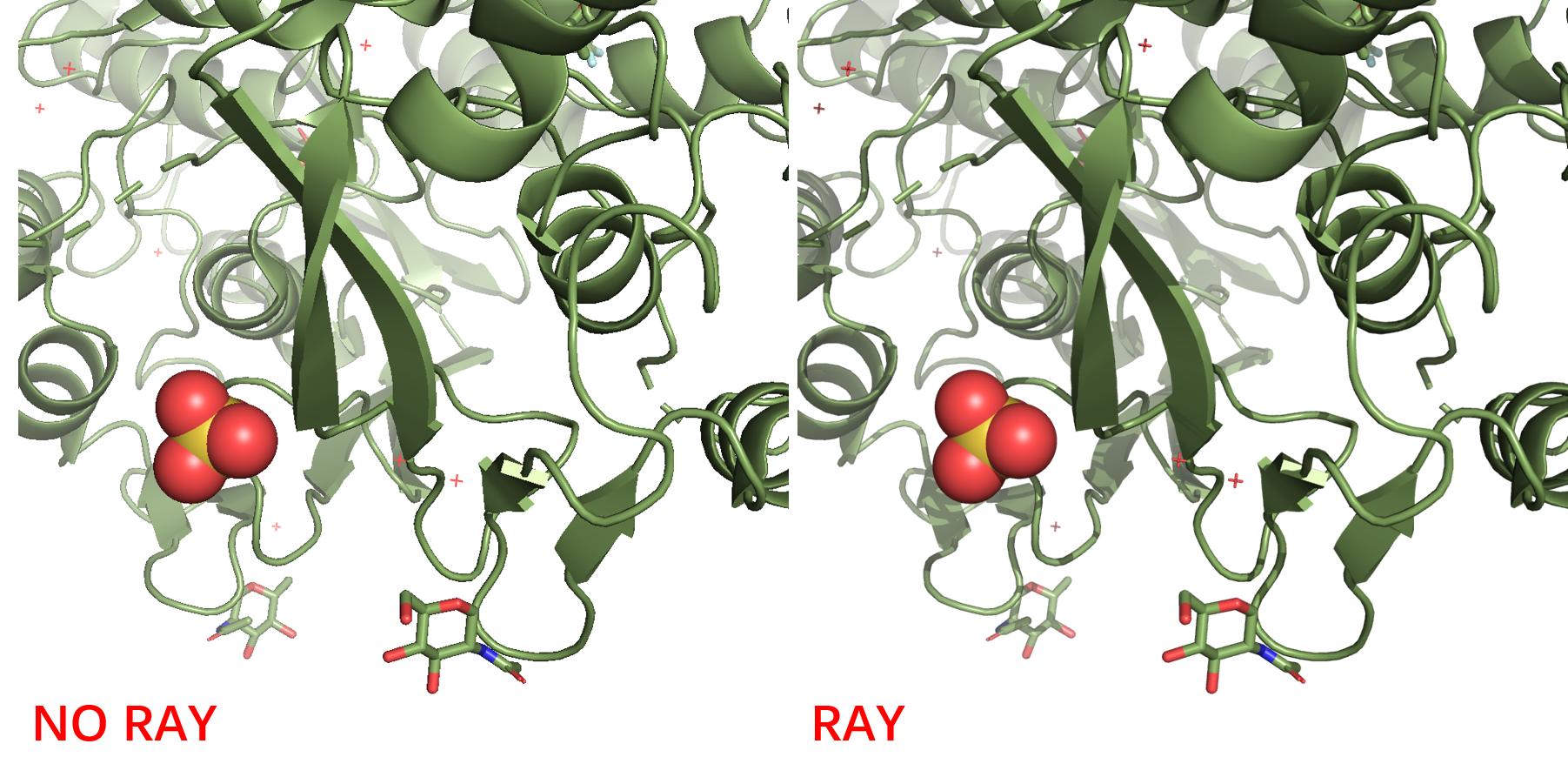
使用 ray 来制作光线追踪的效果,可以使照片更加美观和真实,具体是否使用根据需求选择。
ray [width [,height [,antialias [,angle [,shift [,renderer [,quiet[,async ]]]]]]]]]
width 和 height:含义同上
antialias:抗锯齿,使曲线部分更加平滑
renderer: 渲染器,提供 -1, 0, 1, or 2 四种模式,分别代表 default, built-in,pov-ray, or dry-run,其中 0 是默认模式。
输出图片的背景一般为白色,可以运行 bg_color white 轻松修改。注意仅支持颜色的英文拼写,不支持 16 进制或 RGB 格式的颜色,但是部分支持颜色通过在字母后加数字来修改颜色的透明度,如 grey30 。
下面提供几个示例注意输出图片的区别。
png image1.png, width=1000, height=1000,dpi=300
png image2.png, 1000, 1000,dpi=300
ray
png image3.png, 1000, 1000, dpi=300, ray=1
输出的 3 张图片中 image1 和 image2 完全一样,下图展示 image3 与它们的区别。可以明显看出运行 ray 后输出的图片,具有阴影效果,并且曲线部分更加平滑,同时没有背景,总体感觉右边的比左边的图片更加美观,所以推荐使用。
6.蛋白的展现形式指令(show and as)
使用 show 和 as 均可更改蛋白的外观,其用法也相同。 准确的来说 as 是更改外观,而 show 是展示外观,言外之意就是可以叠加多种形式。
as/show representation [, selection ]
若不知道 as/show 可以填写什么形式,按一下 tab 键会显示全部可以输入的形式;selection 则是填写选择需要改变的部分,其写法非常多,可以在接着前面补全之后,再按 tab 键 一次,感受一下有啥选项,默认为 all。以之前的两个结构为例,依次运行下面指令并观察所产生的变化。
load 6mvd.pdb
as sticks
show cartoon
load 4hbk.cif
reset
show cartoon, 4hbk
7.设置双键显示及小球大小(valence and sphere_scale)
load 4hbk.cif
as sticks
set valence, 0 #关闭双键显示
set valence, 1 #开启双键显示
as sphere
set sphere_scale, 0.5
8.cartoon 模式设置(cartoon)
可以使用 cartoon type, selection 来设置。type 中内置 10 种方案以供选择,其中 selection 参数说明同上。
as cartoon
cartoon loop
cartoon tube
cartoon putty
cartoon automatic
其中 automatic 模式中还内置了三种模式(default、Fancy Helices 和 Cylindrical Helices),运行下面命令观察变化。
# 开启 Fancy 模式
set cartoon_fancy_helices, 1
# 开启 cylindrcal 模式
set cartoon_cylindrical_helices, 1
# 设置圆柱的粗细
set cartoon_helix_radius,0.9
set cartoon_helix_radius,2.5
将 Fancy 和 Cylindrical 关闭(后面参数设置为 0),返回 default 模式。
在 default 模式下还可以设置 α-helix 的长宽。
set cartoon_oval_length, 0.8 # default is 1.20
set cartoon_oval_width, 0.2 # default is 0.25
9.设置颜色和透明度(color and transparency)
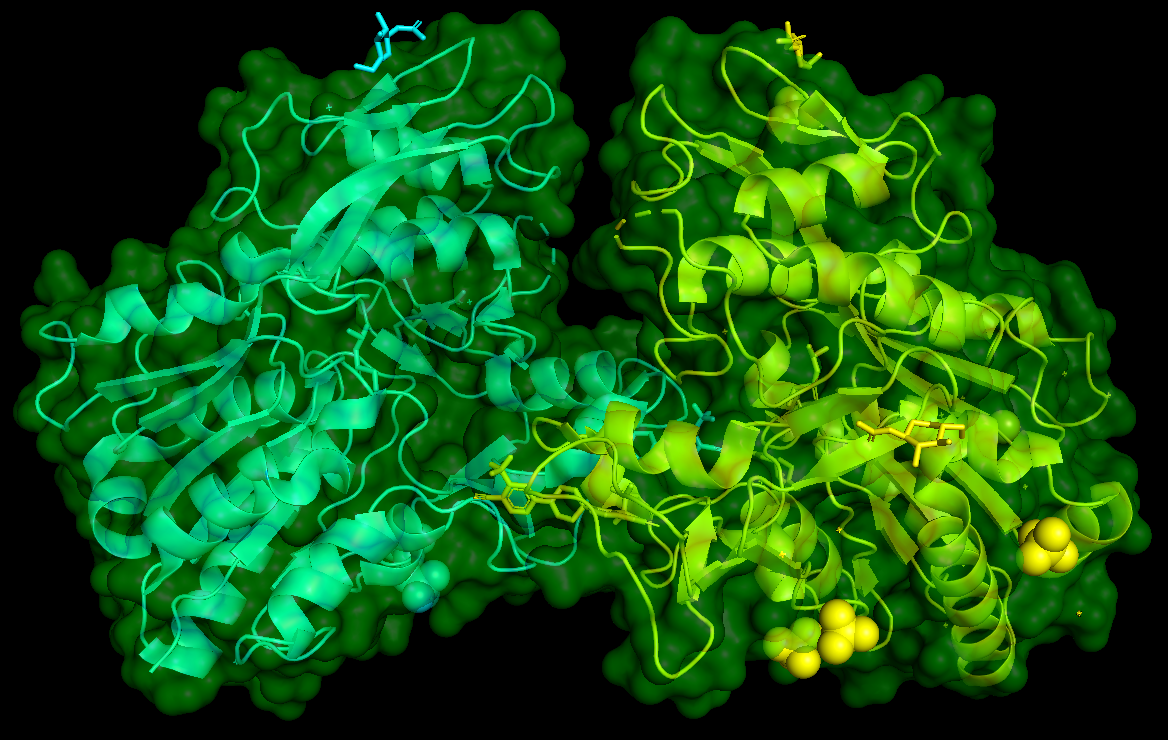
使用 color color [, selection ] 来设置颜色。 而透明度一般会用在 surface 和 cartoon 同时存在的时候置,按照如下指令操作并观察变化,最后通过鼠标旋转可以获得如下图片。
load 6mvd.pdb
color cyan # 修改整体颜色为cyan
color yellow, chain A # 将A链颜色改为yellow
show cartoon #显示cartoon
show surface #显示表面
hide lines #隐藏lines
set surface_color,green #修改surface颜色为green
set transparency,0.6 #设置表面透明度为0.6
set surface_quality,1 #设置表面质量
10.选择命令(select)
使用方法为:select name, selection [, enable [, quiet [, merge [, state [, domain ]]]]]
select chA, chain A # 选择A链,命名为chA
select ( resn HIS ) # 选择HIS
select near142, resi 142 around 5 # 选择142位附近的5个残基,命名为near142
select catoms, elem C # 选择所有的碳原子基,命名为catoms
select protein, (byres polymer & name CA) # 选择蛋白,命名为protein
select nucleic, (byres polymer & name P) # 选择核酸,命名为nucleic
select rna, (byres polymer & name O2) # 选择RNA,命名为rna
select dna, (nucleic & !rna) # 选择DNA,命名为dna
11.查看二硫键及删除配体分子5A以外的水分子
load 6mvd.pdb
show sticks, (cys/ca+cb+sg) and byres (cys/sg and bound_to cys/sg)
load 4hbk.cif
remove resn hoh and (resi 307 gap 5)
12.叠合两个object (super and align)
可以使用super 和 align 进行担保叠合。两者用法基本一致,但针对的叠合的情况有所不同,super 主要基于结构进行叠合,序列相似度较低时效果较好,align 主要基于序列进行叠合,当序列相似性大于 30 % 效果较好。
# super自动叠合4hbk和6mvd
super 4hbk, 6mvd
# 指定4hbk的A链的2-191位残基和6mvd的B链的2-191位残基进行叠合
super 4hbk & resi 2-191 & chain A, 6mvd & resi 2-191 & chain B, object=supeAB
# align自动叠合4hbk和6mvd
align 4hbk, 6mvd
参考资料:
1.PyMOL中文教程
2.PyMOL Command Reference
3.Pymol(1.8.6)作图技巧之cartoon和surface镶嵌模型
