Swiss PdbViewer简要说明
本文主要对 SPDBV 的一些简单用法进行讲解,如有更高需求可以去看官网查看相应说明。
1.Swiss-PdbViewer的下载
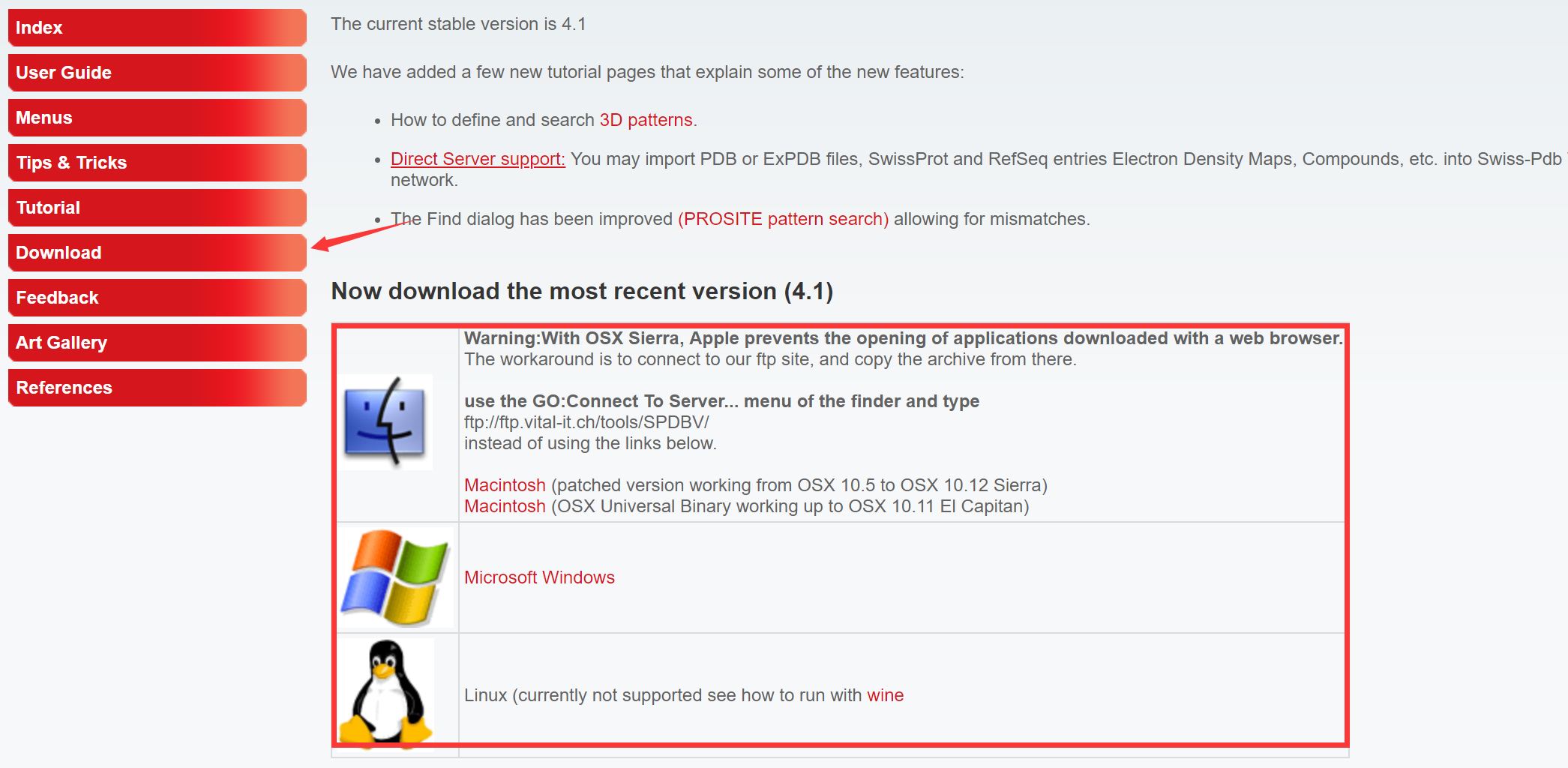
进入 Swiss PDB Viewer 官网,如下图指示来下载对应版本的软件,文件夹中提供下载好的Windows版本软件SPDBV_4.10_PC.zip,解压即用无需安装,双击spdbv.exe即可。
2.文件的导入及保存
2.1 文件的导入
类似于VMDA软件的导入有两种方式:
- 拖拽导入:将pdb文件直接拖拽至打开的程序主界面即可
- 点击导入:点击
File→Open PDB File后,在弹出的对话框中选择需要编辑的pdb文件,快捷键Control + O
2.2 文件的保存
可以保存为多种形式,主要保存pdb和png格式文件。File中的Save下有Current Layer和Image分别保存为pdb和png格式文件。
3.主界面按钮简介
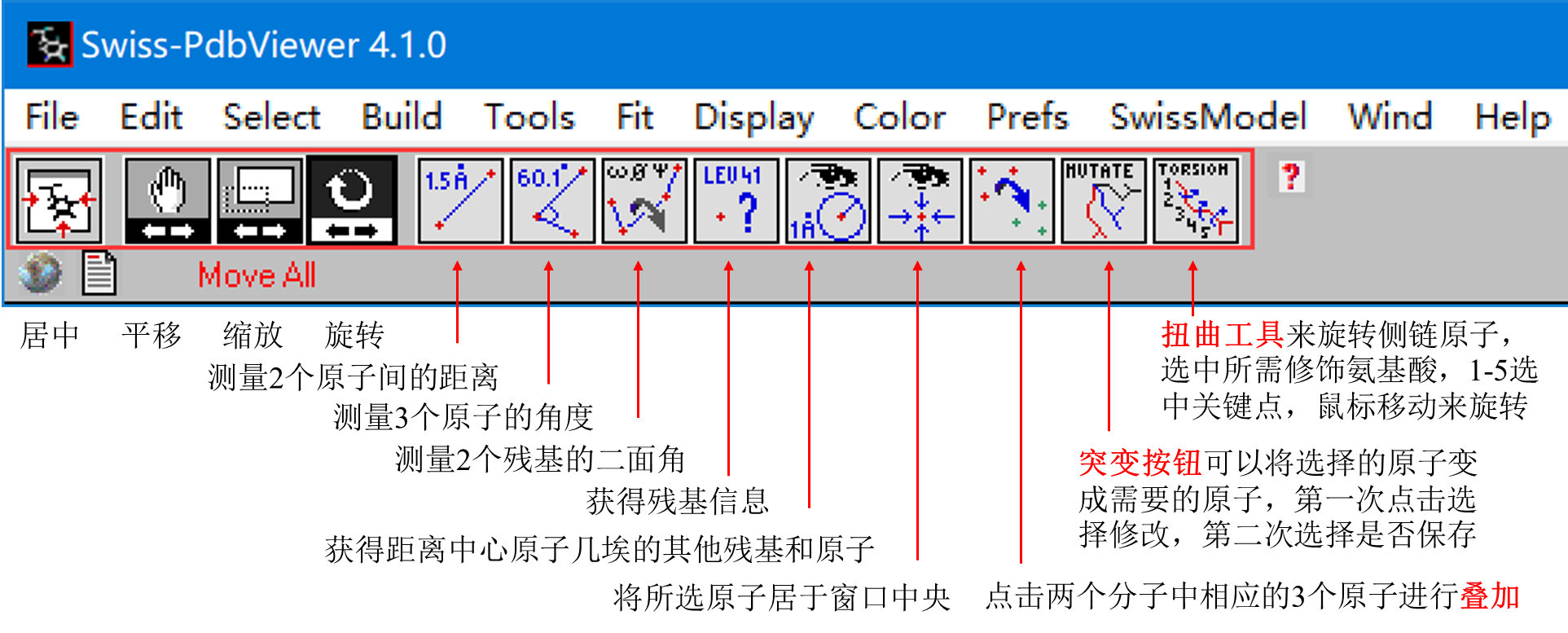
4.Wind中常用窗口简介
4.1 Control Panel
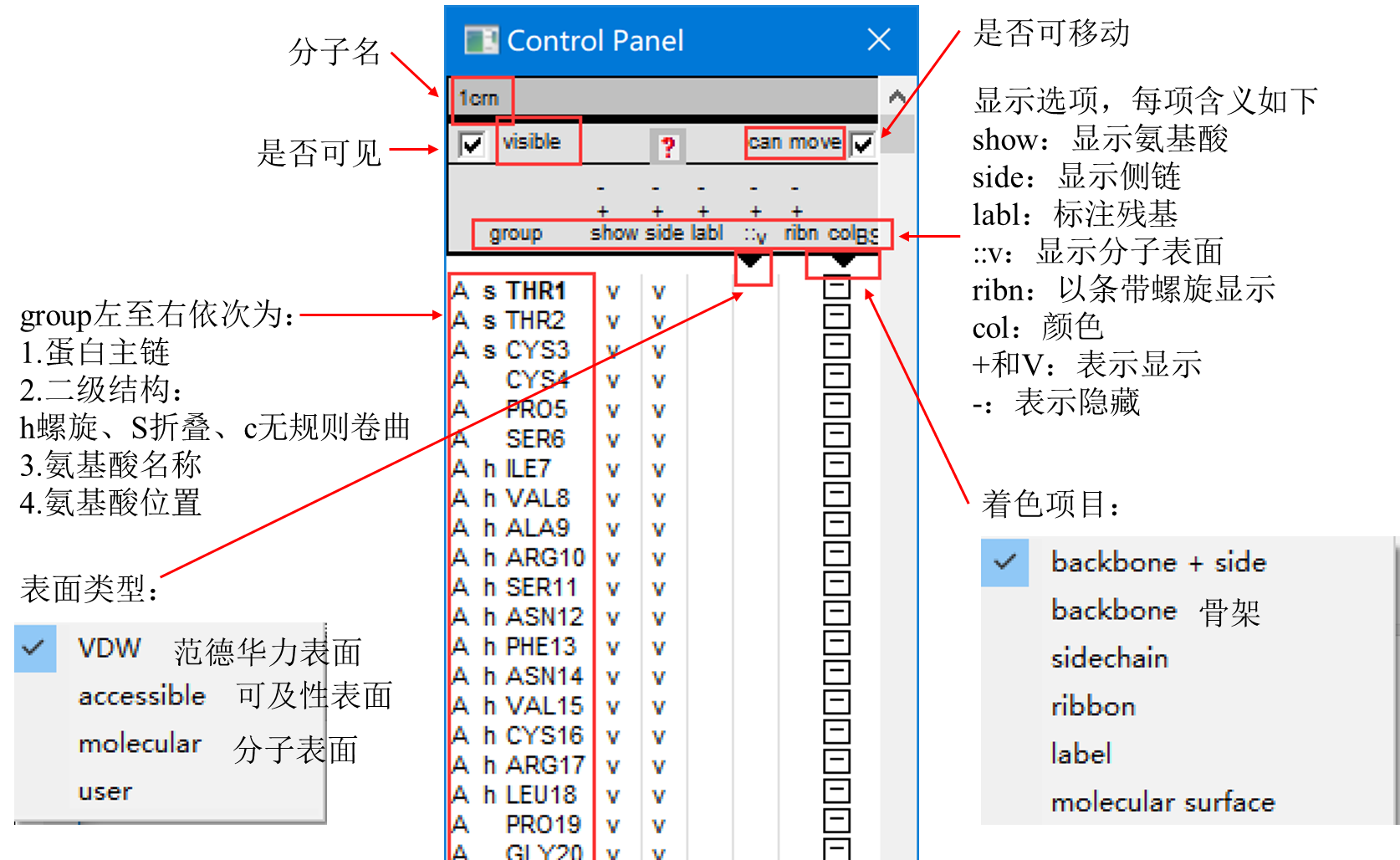
其中每个选项下面的 V 通过鼠标左键点击或左键拖动来选中或移除。通过不同显示效果及颜色标记的组合来达到所需效果。
4.2 Layers Info

其他选项中常见说明如下:
- sel 选中
- grp 分组
- vis 可见
- mov 可移动
- axis 显示坐标轴
- CA 显示α-碳原子
- O 显示骨架氧原子
- H 显示氢原子
- Hbnd 显示氢键
- Hdst 显示氢键间距离
- side 显示侧链
- AlnW 是否出现在比对窗口
- mdl 是否作为同源建模时的模板,星号表示作为模板
- SelGrp 每个分子中当前选中的基团数
4.3 Alignment
点击下图箭头所指按钮可以显示序列比对的详细信息。

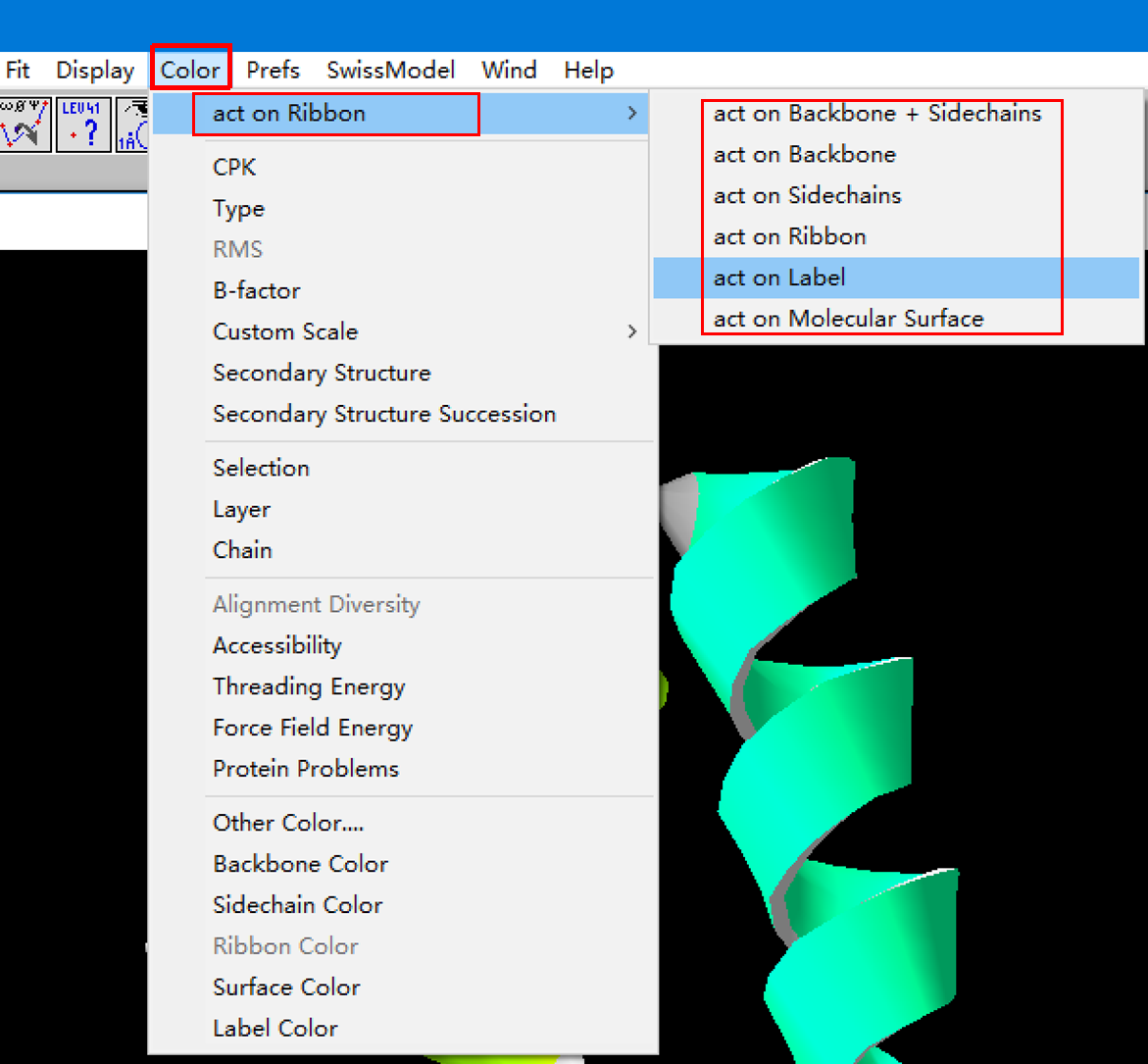
5.Color选项
与Control Panel中类似,需要先选择着色位置再进行着色,color选项中提供了一些常用的选项,可以一键着色比Control Panel中着色要方便不少。建议先在Color中着色后,再通过Control Panel进行微调。
按照下图所示对着色位置进行选择后,再进行后续着色。
5.1 CPK
将所有残基基团颜色恢复到标准状态。
- C原子:白色
- O原子:红色
- N原子:蓝色
- S原子:黄色
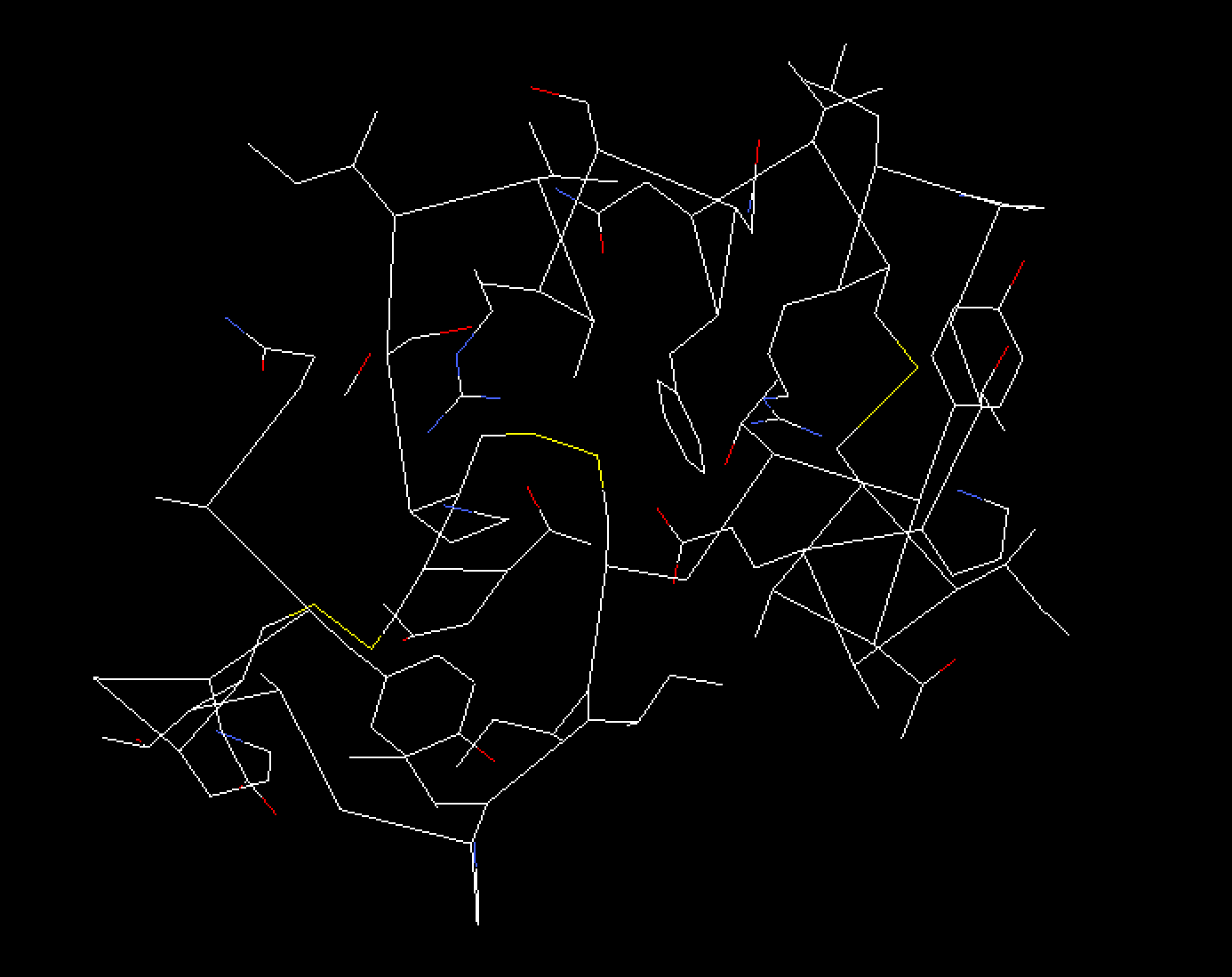
5.2 Type
根据氨基酸残基的化学类型着色。
- 无极性氨基酸:灰色
- 不带电极性氨基酸:黄色
- 带负电的氨基酸:红色
- 带正电的氨基酸:蓝色
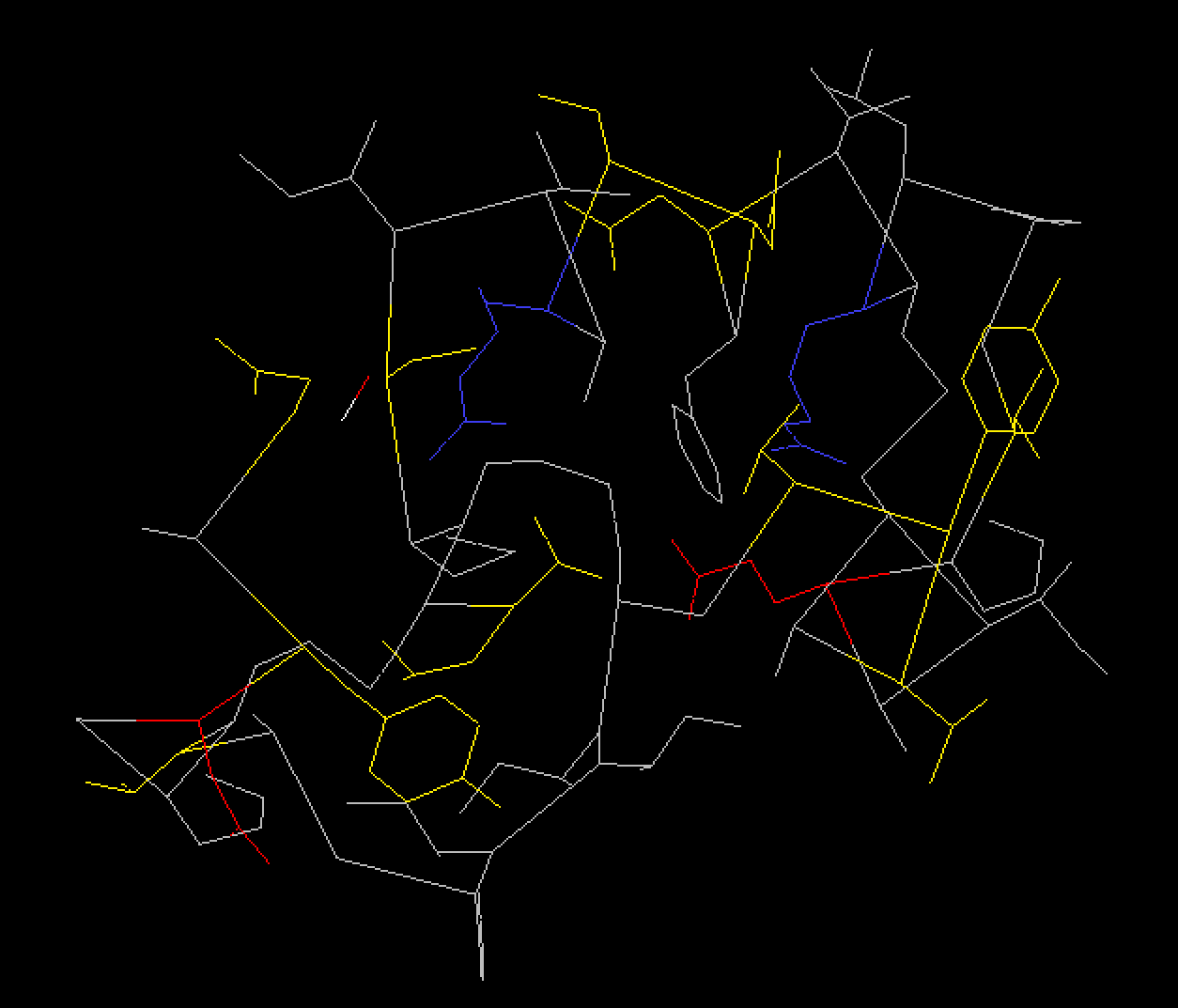
5.3 B-factor
根据B因子(温度因子)着色。B因子对于一个原子来说指的是该原子在一般模型与其他模型的位置间的平均距离,反应分子各部分的摇摆性或活动性。
可以利用B因子来判断其他模型与一般模型额一致性。若所测模型中该原子位置变化不大用深蓝色显示,反之用红色显示。
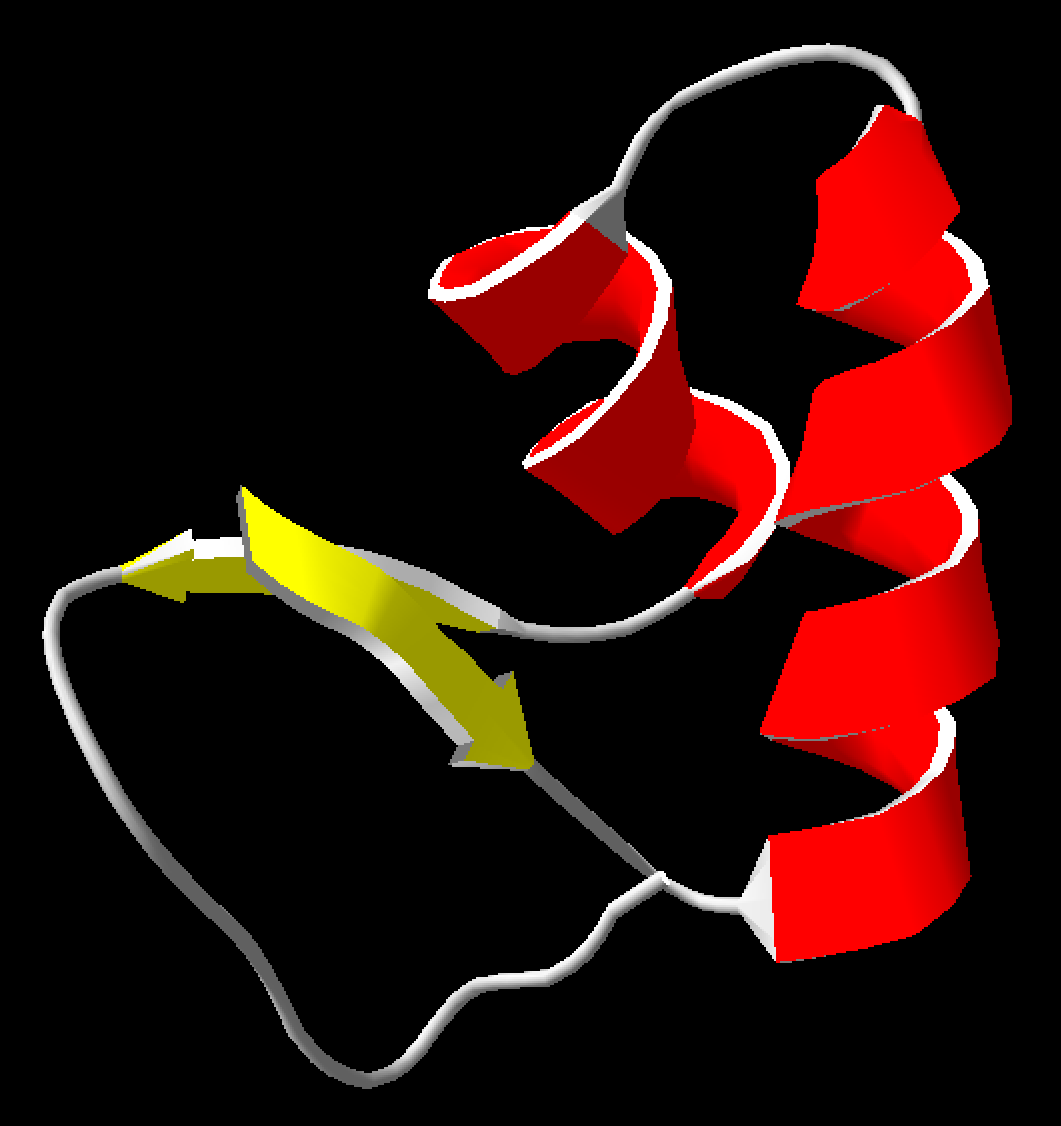
5.4 Secondary Structure
根据不同二级结构着色,同一种二级结构使用相同的颜色。
- 螺旋(helix):红色
- 折叠(strand):黄色
- 无规卷曲(coil):灰色
5.5 Secondary Structure Succession
根据不同的二级结构着色,同一种二级结构也使用不同颜色。
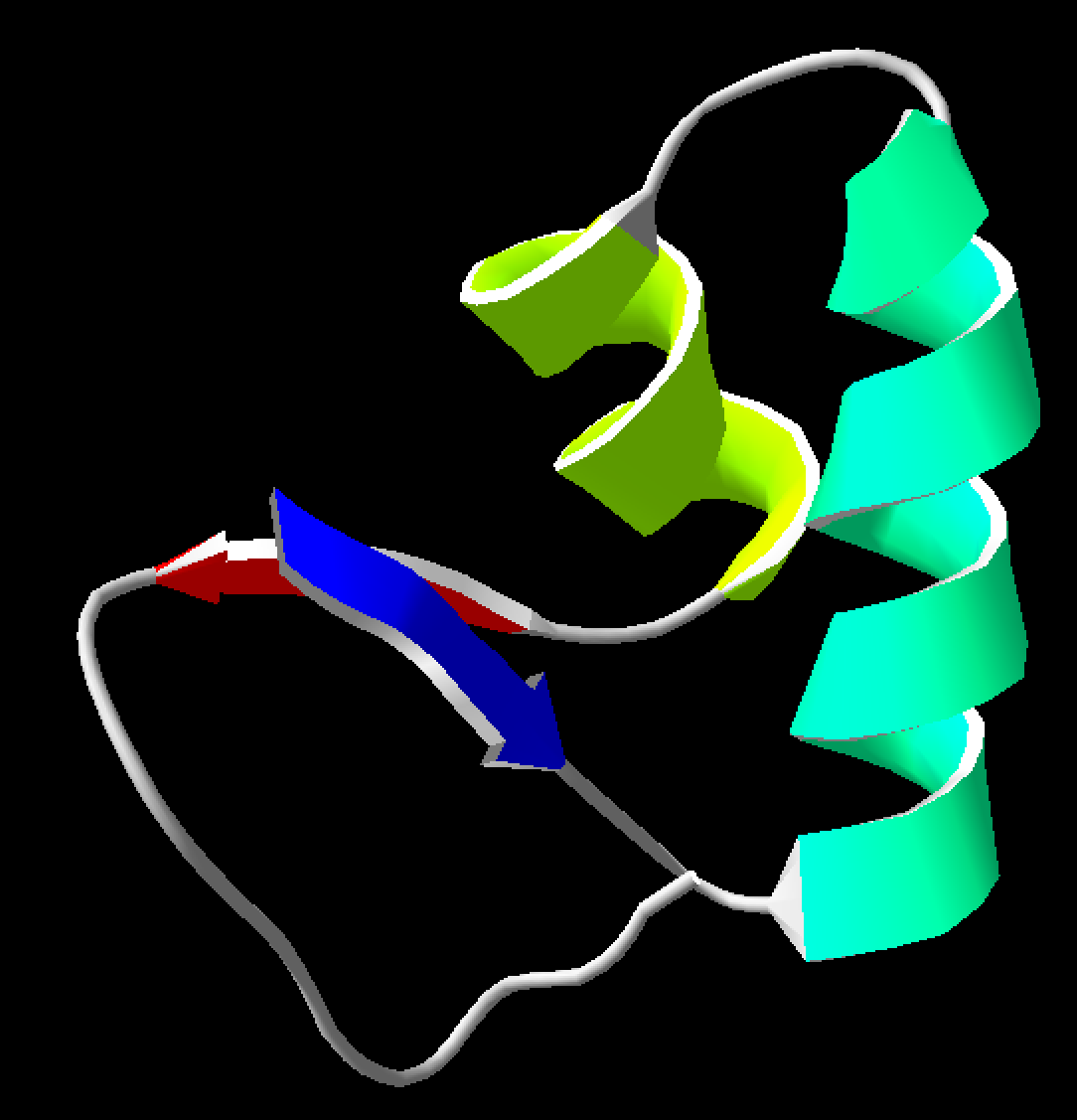
5.6 其他
- Layer:根据图层着色,一个蛋白结构即一个图层,在比较几个蛋白结构的时候使用
- Chain:根据主链着色
- Accessibility:根据每个氨基酸与周围溶剂接触程度着色,接触的少为蓝色,完全暴露在溶剂中为红色
6.比较蛋白结构
两种方式比较来实现不同蛋白三维结构间的拟合:
- 通过主界面中的叠加按钮进行拟合,该操作是自主选择需要一一对应的3个原子来进行拟合
- Fit 菜单中,Magic Fit 选项进行拟合,该选项主要是通过序列信息进行拟合,从而发现差异
参考资料:
swiss_pdb_viewer软件简介.pdf
Swiss-PdbViewer软件的使用.pdf
