ggalt
还在用 PPT 或者 AI 给图画圈圈吗?最近发现这些事情可以用 ggalt 来做,几行代码,解决自己画半天还不满意的情况,可谓是省时省力。
举个实际应用的例子,PCA的结果展示中经常需要将某一类群圈起来,当然不止是这种情况,凡是需要画圈圈的 ggalt 都是可以做的,话不多说直接上代码(主要采用参考资料1中的代码,本文做了一些修改和说明)。
## 安装 ggalt
BiocManager::install("ggalt")
## 加载包
rm(list = ls())
library(ggplot2)
library(ggalt)
library(ggfortify)
library(patchwork)
theme_set(theme_classic())
## 对iris数据集进行PCA,并获得PC坐标,完成数据的准备
df <- iris[c(1, 2, 3, 4)]
pca_mod <- prcomp(df)
df_pc <- data.frame(pca_mod$x, Species=iris$Species)
## p1直接展示PCA的结果
p1 <- ggplot(df_pc, aes(PC1, PC2, col=Species)) +
geom_point(aes(shape=Species), size=2) +
labs(title="Iris Clustering (P1)",
caption="Source: Iris") +
coord_cartesian(xlim = 1.2 * c(min(df_pc$PC1), max(df_pc$PC1)),
ylim = 1.2 * c(min(df_pc$PC2), max(df_pc$PC2))) +
scale_color_manual(values = c('#e41a1c','#377eb8','#4daf4a'))
## p2使用ggalt将三个物种分别圈出
df_pc_set <- df_pc[df_pc$Species == "setosa",]
p2 <- p1 + labs(title="Iris Clustering (P2)") +
geom_encircle(data = df_pc_set, aes(x=PC1, y=PC2), s_shape=0.5, expand=0.05, size=5, show.legend = T) +
geom_encircle(data = df_pc[df_pc$Species == 'versicolor',], s_shape=0.5, expand=0.05, size=5, fill='#377eb840', show.legend = F) +
geom_encircle(data = subset(df_pc, Species=='virginica'), s_shape=1, expand=0.05, fill='#4daf4a40', colour="#4daf4a00")
p1 + p2
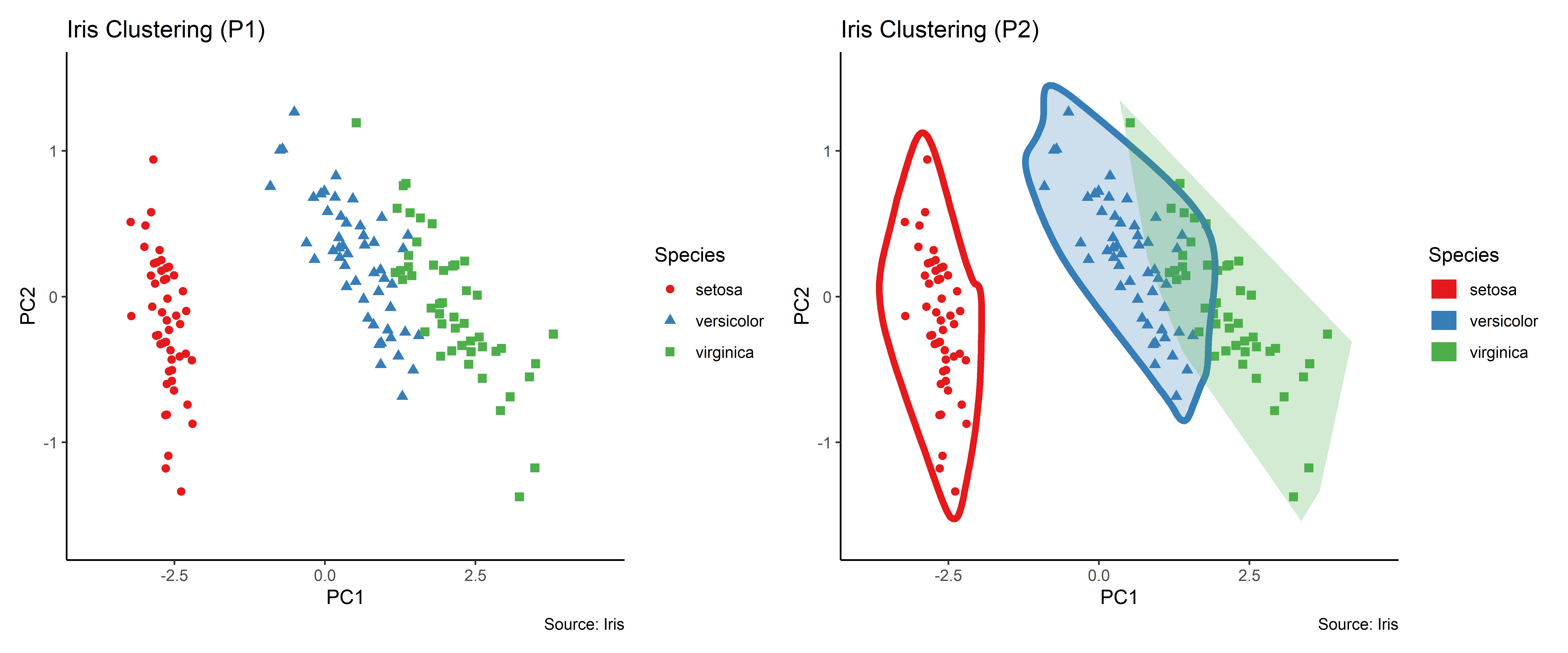
上图的变化利用 ggalt 中的 geom_encircle 函数完成,想要完成上面的各种变化只需要了解下面几个问题即可。
怎么指定所需圈的数据?
若不指定,则会圈出所有数据。
setosa 采用 ggplot2 输入数据的方式,先外部构建 df_pc_set ,然后指定 xy 轴所使用的数据,这里提醒一下, aes(x=PC1, y=PC2) 是可以使用其他列的数据,根据需求改动即可。
versicolor 和 virginica 则分别使用数据框提取子集和使用 subset 函数提取子集的方式指定所需圈出的数据。
画的圈有哪些可调的参数?
圈的平滑度 s_shape:0-2之间的一个数,以1为分界线,向上平滑,向下崎岖,1为直线连接。
圈的拓展度 expand:负数收缩,正数扩张。
圈的粗细 size:注意是圈的线的粗细。
圈的颜色 fill 和colour:二者均为修改颜色的参数,但是分别对应圈内填充的颜色和圈的线的颜色。
圈的图例 show.legend:后接逻辑值(TRUE 或 FALSE),根据所需情况进行选择。
小技巧
上图中并没有使用 alpha 参数来调整透明度是怎么获得透明效果的填充,而且 virginica 圈的线是怎么消失的?
上述问题均通过16进制颜色后面添加一个两位数来调整颜色的透明度,所不同的是完全透明和半透明。而之所以不用 alpha 调整透明度,是因为它只能调整填充色的透明度,不能调整线的透明度,四舍五入就等于我不想用。
最后需要说明的是 ggalt 的功能远不止 画圈圈,有兴趣的可以去参考资料2中继续挖掘。
